Árvores de decisão (Decision Tree), como o próprio nome sugere, é um dos métodos mais utilizados para a tomada de decisão. Essa abordagem é amplamente utilizada em modelos de machine learning, mas também encontra espaço em outras áreas, como a pesquisa operacional.
Existem vários algoritmos que podem ser utilizados nessas árvores, dentre eles o ID3, C4.5 e o CART. O foco será esta última abordagem, que é a utilizada pelo scikit-learn. Ela se baseia na separação binária (True | False) de condições utilizando os atributos — features — dos dados.
Esses modelos de aprendizado supervisionado são empregados em tarefas tanto de regressão quanto classificação. O objetivo do método é criar uma árvore que aprende com os dados por meio de regras básicas, chamadas de if-then rules. O modelo identifica qual é o melhor atributo para separar os dados utilizando o critério escolhido, que, neste caso, pode ser entropy ou gini.
A imagem abaixo ilustra um exemplo geral dessas árvores e alguns de seus conceitos utilizados:
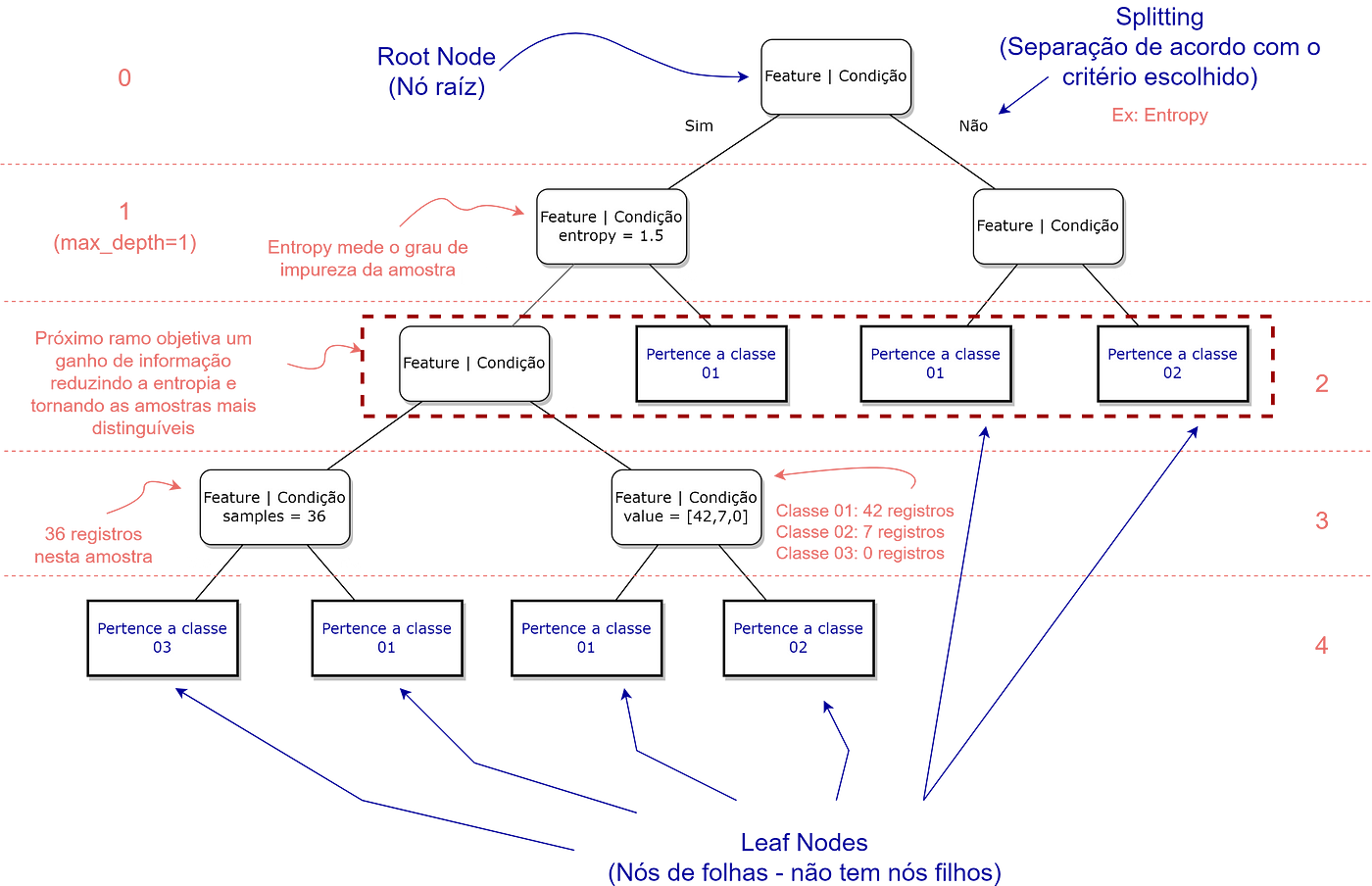
Algumas Vantagens das árvores de decisão:
- É um modelo intuitivo e simples de interpretar. Além disso, as árvores de decisão podem ser visualizadas.
- Não necessita de grandes pré-processamento de dados, como a normalização ou escalonamento de dados.
- Utiliza o modelo conhecido como caixa branca. Ou seja, as decisões que o modelo toma podem ser explicadas e interpretadas.
Desvantagens:
- Pode criar regras muito complexas, que vão gerar overfitting nos dados.
- Podem ser instáveis, então pequenas variações podem gerar modelos de árvore completamente diferentes.
- Para ter predições mais robustas, é aconselhável fazer ensembles.
O objetivo deste artigo é ajudá-lo na compreensão das DecisionTree e avaliar alguns métodos que podem ajudá-lo na interpretação dos seus dados e do seu modelo. Caso tenha interesse, você pode acompanhar o código no Colab.
Neste artigo, iremos utilizar uma base de dados para tarefas de classificação do próprio sklearn, sobre vinhos. Ela tem 178 registros e cerca de treze features.
# importando os dados do toy dataset wine
x,y = load_wine(return_X_y =True, as_frame=True)
# separando em treino e teste
x_train, x_test, y_train, y_test = train_test_split(x,y,random_state=42)Inicialmente, vamos instanciar nossa árvore com todos os parâmetros default. Utilizaremos o clássico fit para treinar o modelo.https://thalesferraz.medium.com/media/7610e1680b4d1f22ee81ea90a3605012.
Já foi falado muitas vezes que esse modelo é muito fácil de interpretar, e, inclusive, podemos visualizá-lo. Chegou a hora de nosso plot!
Para isso, utilizaremos o método do scikit-learn plot_tree!
# criando o fig e o axes
fig, ax = plt.subplots(figsize=(8,6),dpi=92)
#criando o plot
plot_tree(tree,ax=ax)
#plotando o gráfico
plt.tight_layout();
Eu deixei em um tamanho menor para refletirmos: e se houvesse várias features? Ou seja, várias colunas em nosso dataset? Nossa árvore seria gigantesca!
É por conta disso que existem alguns parâmetros que podemos mudar no plot_tree. Por exemplo, ao setar o max_depth como 2, nós limitamos a visão da profundidade da árvore para 2. As demais profundidades ficam simbolizados como três pontos (…).
Se você der uma olhada na primeira linha de cada quadrado (nó), ele exibe um símbolo X [índice]. Este é o jeito que o modelo entende cada feature. Por exemplo, o x[9] é o nono elemento da nossa lista de atributos:
['alcohol', 'malic_acid', 'ash', 'alcalinity_of_ash', 'magnesium',
'total_phenols', 'flavanoids', 'nonflavanoid_phenols',
'proanthocyanins', 'color_intensity', 'hue',
'od280/od315_of_diluted_wines', 'proline']
Ou seja, color_intensity. Por conta disso, para ficar mais legível, podemos acrescentar ao parâmetro feature_names uma lista do nome de nossos atributos. Fazemos isso simplesmente setando feature_names como x_train.columns.
O plot_tree possui muitos outros parâmetros que podem auxiliar na interpretação do modelo. Convido-o a visitar a documentação e a procurar por mais alguns que possam agradá-lo!
No código abaixo, trouxe alguns novos parâmetros e seu plot. Os que estão com # antes não estão sendo considerados no código. Deixei, apenas, caso alguém tenha interesse. Do lado de cada parâmetro, deixei uma explicação simples do seu efeito.
# criando o fig e o axes
fig, ax = plt.subplots(figsize=(12,8),dpi=92)
#criando o plot
plot_tree(tree, # a decision tree que será plotada
feature_names = x_train.columns, # trará o nome das features utilizadas na primeira linha de cada nó
ax=ax, # plotamos no axes criado do matplotlib
precision=1, # precisão dos valores numéricos
max_depth=2, # escolhemos a profundidade da árvore
# proportion = True # retorna a proporção dos valores das amostras
#fontsize = 20 # mudar o tamanho da fonte
)
#plotando o gráfico
plt.tight_layout();
Show! Acredito que até agora tudo tenha ficado bem claro.
Porém, uma dúvida pode surgir: como as árvores de decisão fazem a separação considerando o atributo? Por exemplo, de onde ela tirou que color_intensity <= 3.8 é um bom splitting?
Para isso, ela se baseia nos hiperparâmetros das DecisionTrees: criterion, que é o gini por default, e splitter, que é best por default. Ou seja, ela faz a separação buscando reduzir o coeficiente de gini da melhor (best) forma possível.
Utilizando a entropy , o objetivo vai ser aumentar o ganho de informação reduzindo a entropia da próxima profundidade.
Obs.: O coeficiente de gini ou entropy mede a heterogeneidade dos dados. Esta separação busca reduzir esta impureza no próximo ramo (próxima profundidade) como um todo.
Se voltarmos a dar uma olhada em nossas árvores plotadas, vamos observar que ela exibe outros termos: samples e value:
- samples: é a quantidade de registros daquela amostra.
- value: quantos registros tem de cada classe naquela amostra.
A DecisionTreeClassifier tem muitos outros hiperparâmetros que podem auxiliar em uma melhor classificação de seu modelo. Novamente, convido-o a visitar a documentação e a procurar por mais alguns que possam ajudá-lo!
No código abaixo, trouxe alguns novos hiperparâmetros. Do lado de cada hiperparâmetro, deixei uma explicação simples do seu efeito no modelo.
tree = DecisionTreeClassifier(
criterion='gini', # gini vem por default, mas podemos optar por entropy
splitter='best', # a estratégia utilizada para fazer a separação de cada nó
# ela também pode ser feita de forma randômica utilizando 'random'
max_depth= None, # a máxima profundida que sua árvore de decisão pode ter
# se for None ela vai buscar a máxima pureza possível
min_samples_split = 2, # o mínimo de registros necessários para que uma separação seja feita
min_samples_leaf = 1, # o mínimo de registros necessários em cada nós-folha (veja a primeira imagem)
max_features = None, # o número de atributos que será considerado durante o split
# None -> seleciona todos os atributos, 'sqrt' -> raiz quadrada do número dos atributos, 'log2' -> log de base 2 do número de atributos
max_leaf_nodes=None, # a quantidade máxima de nós-folha que a árvore pode ter
# se for None ele não limitará o número de nós-folha
min_impurity_decrease=0.0, # o split irá ocorrer em cada nó se o decréscimo da impureza foi maior ou igual a este valor
random_state= 42, # permite o notebook ser reproduzível
# veja mais parâmetros na documentação oficial!
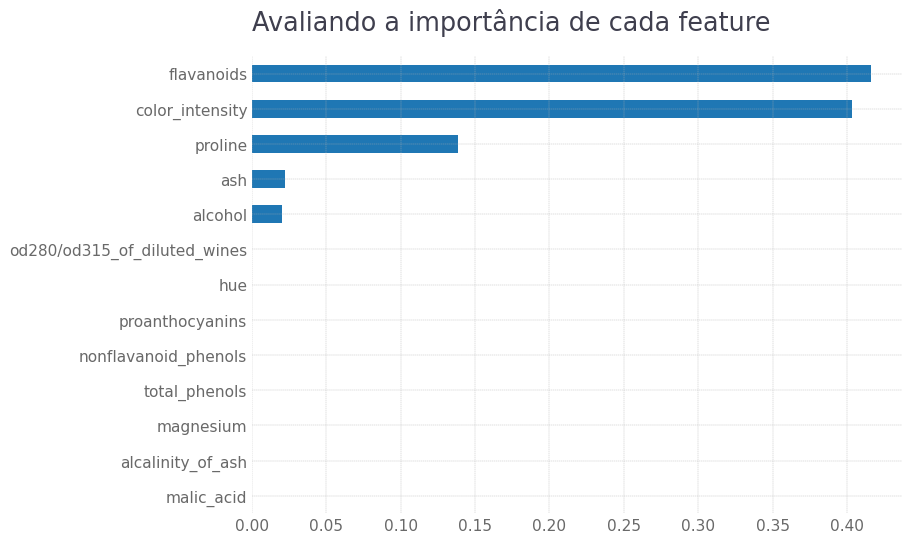
)Outro método interessante das árvores de decisão é o feature_importances_. Este método traz um score de quais atributos melhor ajudaram o modelo a aprender sobre os dados, baseando-se na redução de impureza trazida por cada atributo.
Para calcular este score, o scikit-learn utiliza o gini importance (este vídeo do Machine Learning Mastery explica detalhadamente os cálculos realizados).
É importante ressaltar uma resposta dada por Gilles Louppe, um dos desenvolvedores do scikit-learn, no stack overflow: há muitas maneiras de se calcular a feature importances , não existe um consenso sobre isso. No scikit-learn, optou-se por utilizar o gini importance.
#criando um dicionário com as variáveis
dic = {'score':tree.feature_importances_,'features':x_train.columns}
#criando um dataframe com os dados
df = pd.DataFrame(dic).sort_values('score',ascending=False)
df
E se quisermos exportar nossa árvore de decisão para outras pessoas?
Geralmente, quando utilizamos este tipo de template (com setas, nós etc.), salvamos o arquivo no formato DOT. Conseguimos realizar essa tarefa através do método export_graphviz.
O código abaixo reproduz esse processo:
# exportando uma árvore de decisão em DOT format
export_graphviz(tree, # árvore de decisão a ser exportada
out_file = 'arvore.dot', # nome do arquivo output
feature_names = x_train.columns, # retornar com as features
precision=2, # número de digitos da precisão
filled = True, # pinta os nós de acordo com cada classe
class_names=['class_0','class_1','class_2']) # lista do nome das classes
#special_characters=False, # quando False ignora os caracteres especiais
#label = 'root', # as informações das features aparecem apenas na raíz
#leaves_parallel = True) # faz com que as folhas fiquem paralelas
#rotate = True); # retorna a árvore na horizontal
Além dos formatos já apresentados, ainda é possível representar as regras de nossa árvore de decisão através de texto, utilizando o módulo export_text.
print(export_text(tree, # árvore de decisão treinada
show_weights=True, # retorna o número de amostras de cada classe
spacing=5)) # dá um espaçamento entre os nós|----- feature_9 <= 3.82
|----- feature_12 <= 1010.00
| |----- feature_2 <= 3.07
| | |----- weights: [0.00, 46.00, 0.00] class: 1
| |----- feature_2 > 3.07
| | |----- weights: [1.00, 0.00, 0.00] class: 0
|----- feature_12 > 1010.00
| |----- weights: [1.00, 0.00, 0.00] class: 0
|----- feature_9 > 3.82
|----- feature_6 <= 1.40
| |----- weights: [0.00, 0.00, 36.00] class: 2
|----- feature_6 > 1.40
| |----- feature_12 <= 724.50
| | |----- feature_0 <= 13.14
| | | |----- weights: [0.00, 7.00, 0.00] class: 1
| | |----- feature_0 > 13.14
| | | |----- weights: [1.00, 0.00, 0.00] class: 0
| |----- feature_12 > 724.50
| | |----- weights: [41.00, 0.00, 0.00] class: 0
Em resumo, era isso. As árvores de decisão são muito utilizadas e pouco compreendidas. Espero que este artigo tenha, vamos dizer assim, clareado algumas ideias e gerado insights para alguns de seus projetos!
Caso tenha algum comentário, dúvida ou sugestão, não hesite em me procurar no LinkedIn. Além disso, você também pode procurar alguns de meus projetos de Data Science no Github.





















Pode me tirar uma dúvida, por favor?
O nó raiz no scikit-learn gera sempre apenas 2 ramos, um para a esquerda e outro para a direita, não importando a quantidade de instâncias que haja na variável escolhida. Como e porque ele faz a divisão em apenas 2 grupos? Pergunto isso pelo fato de que, ao estudarmos os algoritmos (entropia e gini), os ramos sempre são na quantidade de instâncias existentes.
Obrigado pela atenção!